|
Wildkatzen haben weder Baue wie etwa Dachs oder Fuchs, noch lassen sich ihre Reviermarkierungen leicht erfassen. Sie sind leicht mit wildfarbenen Hauskatzen zu verwechseln, was Sichtungen oder Fotonachweise erschweren. Gleiches gilt für Feldzeichen (z. B. Spuren oder Kot). Sie lebend zu fangen ist schwierig und in größerem Umfang auch zu aufwändig. (vgl. Weber 2008, S. 4). Deshalb musste eine effektive, neue Methode zum Nachweis entwickelt werden.
Bei der von Karsten Hupe in Deutschland erstmals erprobten Lockstockmethode (HUPE 2004) (basierend auf JOHN L. WEAVER ET AL. 2003; MOWAT & PAETKAU 2002; ZIELINSKI & KUCERA 1995) werden mit Baldrian präparierte Lockstäbe aus rauem Holz aufgestellt, an denen sich die Katzen reiben. Die dabei anhaftenden Haare werden regelmäßig abgesammelt und anschließend genetisch untersucht.
In den Laboren der Naturschutzgenetik des Forschungsinstitutes Senckenberg Gelnhausen werden die Proben weiter bearbeitet. Es kommen zur genetischen Bestimmung von Wildkatzen zwei Verfahren zur Anwendung:
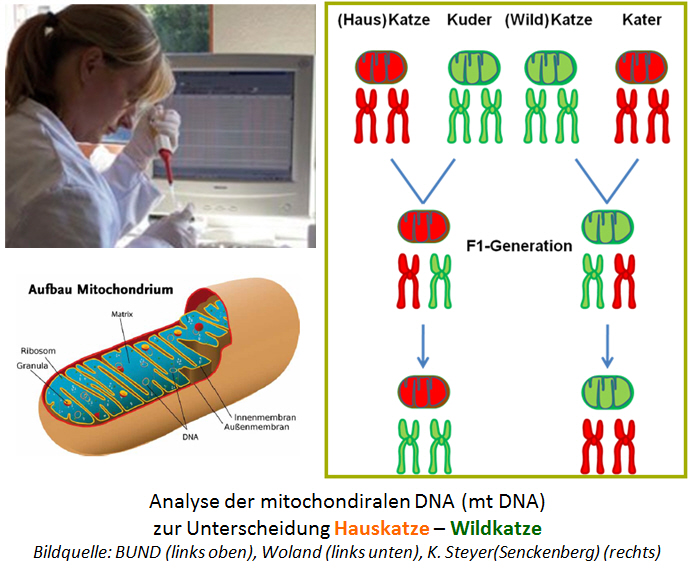
Mit der Herauslösung der mitochondrialen DNA (mt DNA-Analysen bestimmen die Tierart) wird zunächst ein bestimmter Abschnitt der DNA mittels chemischer Verfahren vervielfältigt und danach analysiert. Die Analyse erfolgt durch den Vergleich der DNA-Sequenzen bekannter Wildkatzenproben mit der vorliegenden Probe. Der Vorteil ist, dass viele Mitochondrien in einer Zelle vorhanden und schon ein bis zwei Haare für diese Methode ausreichend sind. Die mtDNA wird von der Mutter vererbt. Blendlinge mit Wildkatzenmutter werden deswegen als Wildkatzen erkannt, solche mit Hauskatzenmutter als Hauskatzen.
Benutzt man Kern-DNA kann man diesen Fehler zwar vermeiden, allerdings wird es kostspieliger, denn man braucht fünf anstelle von einem oder zwei Haaren, da die Kern-DNA seltener vorhanden ist. Abschnitte der Zellkern-DNA, in denen ein kurzer Teil des DNA-Stranges hintereinander vorliegen, werden analysiert (Mikrosatellitenanalyse). Diese DNA Marker sind kurze nicht kodierende DNA Sequenzen, die oft wiederkehren und sich nur auf der Kern-DNA an bestimmten Loci (Auffindort einer Sequenz) befinden. Neben der Bestimmung auf Artniveau kann auch die Analyse des Geschlechts vorgenommen werden. Es besteht sogar die Möglichkeit, nah verwandte Wildkatzen so voneinander zu unterscheiden. Von Individuum zu Individuum können sich die Sequenzen in ihrer Größe (in Basenpaaren) unterscheiden. Diese Größenunterschiede an den 14 verschiedenen Loci ergeben ein Muster, das für jedes Individuum einzigartig ist. So lassen sich auch Wildkatzenpopulationen voneinander abgrenzen (vgl. Weber 2008, S. 5f). Im Rahmen dieses Projektes wurden zunächst mtDNA-Analysen und bei positiver Wildkatzenbestätigung Kern-DNA-Analysen durchgeführt. Hier finden Sie die Ergebnisse der gemeinsamen Lockstockuntersuchungen.